Lasso 分析
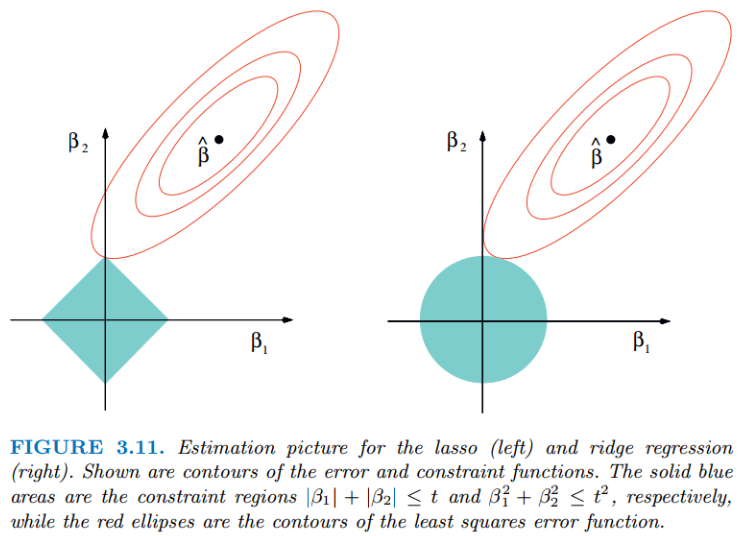
Lasso 回归分析1.原理 LASSO,全称Least absolute shrinkage and selection operator,是一种数据挖掘方法,即在常用的多元线性回归中,添加惩罚函数,不断压缩系数,从而达到精简模型的目的,以避免共线性和过拟合。当系数为0时,同时达到筛选变量的效果。
LASSO回归高效解决了筛选变量的难题:区别于传统的逐步回归stepwise前进、后退变量筛选方法,LASSO回归可以利用较少样本量,高效筛选较多变量。比如在基因组学、影像学、以及其他小样本分析中,LASSO回归都可以派上大用场。
2.实战 R代码添加Times New Roman的字体格式
123456789101112131415161718192021222324library(showtext)library(sysfonts)font_families() showtext_auto()path <- function(x) { font_path <- 'C:/Windows/Fonts/' paste0(font_path, ...
How to use the Hexo codes
Blogs
上述在本地文件夹blogs下打开git bash,后进行相关处理,部署是在github上
Video:Hexo部署使用
Website:博客资料
1.常见的hexo使用命令123456789101112131415161718192021##生成npx hexo g##上传npx hexo d##预览npx hexo serve##新建文章npx hexo n ‘myblog’#使用VPN时,调整Git代理设置git config --global http.proxygit config --global https.proxy#未使用VPN时,取消Git代理设置git config --global --unset http.proxygit config --global --unset https.proxy
MRZhou simplify the mendelian randomization analysis
MRZhouThis R package is designed for the convenience of medical professionals, aiming to further simplify the analysis methods of Mendelian randomization (MR). It is integrated based on the TwosampleMR, MRpresso, and coloc packages. Therefore, whether it is the online data of the IEU GWAS database or local GWAS data (such as FinnGen database), this package can be conveniently executed. This package also includes: GSMR analysis, SMR analysis methods, etc. TWAS/Fusion analysis function method ...
SMR 分析
SMR 教程官方文档:SMR | Yang Lab (westlake.edu.cn)
1.数据获取1.1eqtl数据qtl数据获取主要从GTEx数据库,可以得到各种组织的数据。GTEx数据库的速度很慢建议从SMR官网的数据库下载(华人做的)SMR | Yang Lab (westlake.edu.cn)里面有各种qtl的数据集,下得很快!
eqtl还有血液的数据,样本量很大eQTLGen - cis-eQTLs
直接下载besd二进制格式即可,后期数据可以用smr软件自提
1.2GWAS数据GWAS数据获取的方法很多可以直接去IEU OpenGWAS project (mrcieu.ac.uk)下载,也可以通过R包TwoSampleMR直接从网站上拽。available_outcomes()函数可以看到openGWAS里面所有的gwas数据
当然还有很多别的获取手段。也可以从finngen的GWAS获取
1.3参考基因组用R包下载Download 1000G — download_1000G • bigsnpr (privefl.github.io)
也可以直接链接:http://f ...
LDSC 分析
连锁不平衡分数回归 LD score regression -LDSCLDSC原理为什么要做 LD score regression
在GWAS研究中,多基因性(polygenicity,即若干较小的基因效应)和干扰因素引起的偏差(如隐性关联 cryptic relatedness,群体分层population stratification等)都会造成检验的统计量的分布偏高(inflated)。但我们并不能分辨偏高的统计量到底是来自多基因性还是干扰因素,所以通过LD score regression,我们可以通过研究检验统计量与连锁不平衡(linkage disequilibrium)之间的关系来定量分析每部分的影响。
LDscore的原理?
GWAS检验中,对一个SNP效应量的估计通常也会包含与该SNP成LD的其他SNP的效应,也就是说一个与其他SNP成高LD的SNP,通常也会有更高的卡方检验量。
接下来简单介绍如何进行LDscore regression。使用的软件为ldsc,可以从作者的github中拉取。ldsc为python脚本,clone了ldsc的库之后我们还需要利用an ...
TWAS 分析
TWAS FUSIONTWAS / FUSION (gusevlab.org)
Installation
Download and unpack the FUSION software package from github:
123wget https://github.com/gusevlab/fusion_twas/archive/master.zipunzip master.zipcd fusion_twas-master
Download and unpack the (1000 Genomes) LD reference data:
12wget https://data.broadinstitute.org/alkesgroup/FUSION/LDREF.tar.bz2tar xjvf LDREF.tar.bz2
Download and unpack the plink2R library (by Gad Abraham):
12wget https://github.com/gabraham/plink2R/archive/master.zip ...